Aktuelle Projekte
Entwicklung eines molekularen Krans basierend auf dem Flavoprotein Dodecin
Gefördert durch den European Research Council, ERC
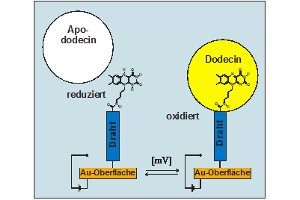
Das riboflavinbindende Protein Dodecin von
Halobacterium salinarum bindet natürliche und
artifizielle Flavine im oxidierten Zustand; Reduktion
führt zur Dissoziation des Holoproteins in Apododecin
und freie Flavine. Basierend auf diesem einzigartigen
Bindungsverhalten soll ein molekularer Kran entwickelt
werden, der durch ein angelegtes elektrisches Potential
geschaltet werden kann. Hierfür soll ein Flavin über
einen molekularen Draht an die elektrisch leitende
Spitze eines Rasterkraftmikroskops (AFM) angebunden
werden. Diese kann als Greifarm eines molekularen Krans
dienen (siehe unten).
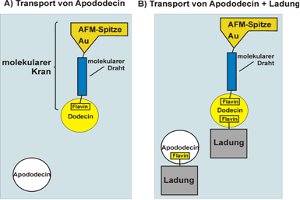
In Abhängigkeit vom angelegten Potential können
einzelne Moleküle Dodecin gebunden und an anderer
Stelle wieder abgelegt werden. Da Dodecin insgesamt
sechs Flavinbindungstaschen aufweist, könnten auch
weitere Lasten angebunden und transportiert werden. Ein
wichtiger Aspekt ist die Auswahl geeigneter molekularer
Drähte zur Anknüpfung von Flavinen an die Spitze eines
AFMs. Hierfür könnten z. B. organische ?
Elektronensysteme oder einwandige Kohlenstoffnanoröhren
dienen. Um diese Moleküle im Hinblick auf eine
Verwendung als molekularer Draht zu untersuchen und
Protokolle zur Anknüpfung an Elektroden zu entwickeln,
sollen zunächst ganze Oberflächen modifiziert werden.
An diese flavinmodifizierten Oberflächen soll dann
Apododecin in Abhängigkeit vom angelegten Potential
angebunden und wieder freigesetzt werden.
Anschließend ist die schrittweise Miniaturisierung
dieser modifizierten Oberflächen bis hin zum
Einzelmolekülniveau unter Verwendung einer AFM-Spitze
als Arbeitselektrode bzw. als Greifarm eines
molekularen Krans geplant. Geeignete Protokolle zur
Oberflächenmodifizierung sollen entwickelt werden und
die modifizierten Oberflächen sollen durch eine
Kombination aus AFM,
Oberflächenplasmonenresonanzspektroskopie, (surface
plasmon resonance spectroscopy, SPR), Ellipsometrie,
Fluoreszenzmikroskopie, Messungen mit der
Quartzkristallmikrowaage (quartz crystal microbalance,
QCM) und elektrochemischen Methoden untersucht werden.
Die dabei gewonnenen Erkenntnisse können von generellem
Interesse für die Entwicklung molekularer Schalter,
Transportsysteme und elektrochemisch aktiver
Oberflächen z. B. für eine Verwendung in
amperometrischen Biosensoren und Biobrennstoffzellen
sein.
Untersuchung von Elektronentransferprozessen in DNA und Entwicklung eines elektrochemischen DNA-Sensors
Gefördert durch das Land NRW
Bei dem Versuch, doppelsträngige DNA (ds-DNA) mit 20
Basenpaaren als molekularen Draht zur Anknüpfung von
Flavinen an Goldoberflächen in einer elektrochemisch
ansprechbaren Weise zu verwenden, hat sich in
vorangegangenen Arbeiten gezeigt, dass die
Leitfähigkeit von DNA hierzu nicht ausreicht (M.
Grininger and G. Nöll, S. Trawöger, E.-K. Sinner, and
D. Oesterhelt; Biointerphases, 2008, 3, 51-58).
Untenstehend ist die Basensequenz der dabei verwendeten
DNA-Einzelstränge sowie die Anknüpfung des Flavins und
des Thiols (geschützt als Disulfid), das Adsorption auf
Goldoberflächen ermöglicht, an die DNA-Einzelstränge
gezeigt.

Andererseits ist aber bekannt, dass reduktiver
Elektronentransfer (ET) durch ds-DNA über große
Distanzen prinzipiell möglich ist. Daher soll
systematisch untersucht werden, wie ET durch
DNA-Monolagen optimiert werden kann und ob sich daraus
die Möglichkeit ergibt, einen Sensor zum Aufspüren von
DNA-Basenfehlpaarungen zu entwickeln. Als Redoxzentrum
soll dabei ein Flavin dienen, das kovalent an einen der
DNA-Einzelstränge angebunden wird. Von Bedeutung ist
dabei die Frage, welche Rolle die Verknüpfung zwischen
DNA und Flavin bzw. zwischen DNA und der
Elektrodenoberfläche spielt. Insbesondere soll die
Länge des Linkers, der die Thiolgruppe zur Adsorption
der DNA auf Gold trägt, variiert werden. Die
Thiolgruppe soll entweder am 3’- oder am 5’-Ende des
gleichen oder des entgegengesetzten DNA-Strangs
angebunden werden, der die Flavinmodifikation trägt.
Außerdem sollen unterschiedliche
Beschichtungsprotokolle erprobt werden, um eine für
eine Sensoranwendung optimale Oberflächenarchitektur zu
erreichen. Durch Kombination von Impedanzspektroskopie
und Oberflächenplasmonenresonanzspektroskopie (surface
plasmon resonance spectroscopy, SPR), soll der
schrittweise Aufbau flavinmodifizierter DNA-Monolagen
charakterisiert werden. Anschließend soll durch
elektrochemische Messungen untersucht werden, wie gut
ET durch die DNA-Monolagen möglich ist und welche
Geschwindigkeitskonstanten dabei erreicht werden
können. Ergänzend zu den elektrochemischen Messungen
soll das Flavoprotein Dodecin, das Flavine nur im
oxidierten Zustand bindet, als Sonde verwendet werden,
durch die eine Änderung des Flavinredoxzustandes
äußerst empfindlich nachgewiesen werden kann. Sollte
sich herausstellen, dass ds-DNA mit 20 Basenpaaren für
effektiven ET zu lang ist, kann auch mit kürzeren
DNA/PNA Doppelsträngen gearbeitet werden (PNA = peptide
nucleic acid). Im Folgenden sind zwei mögliche
Elektrodenarchitekturen zum Aufspüren von
DNA-Fehlpaarungen gezeigt:
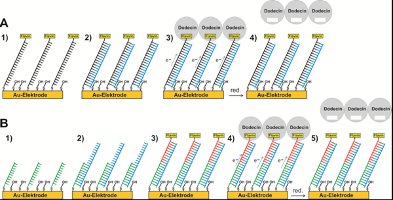
Die Sensorarchitektur in (A) besteht aus einem auf Gold
adsorbierten flavin- und thiolmodifizierten
DNA-Einzelstrang mit 20 Basen (1). Zusätzlich wurde ein
Merkaptoalkohol adsorbiert, um unspezifisch adsorbierte
DNA zu verdrängen und die flavinmodifizierte DNA weg
von der Oberfläche in Richtung Lösung auszurichten.
Dabei muss nicht nur die Belegungsdichte optimiert
werden, sondern es ist auch darauf zu achten, dass die
Länge des verwendeten Merkaptoalkohols eine mögliche
DNA-Hybridisierung nicht behindert. Erst wenn
vollständige Hybridisierung mit dem zu analysierenden
DNS-Einzelstrang erfolgt ist (2), sollte die
elektrochemische Reduktion des Flavins bei einem
vorgegebenen Potential durch ET entlang der Basenpaare
möglich sein. In diesem Fall sollte es auch möglich
sein, Apododecin an das oxidierte Flavin anzubinden (3)
und durch Flavinreduktion auch wieder freizusetzen (4).
Da einzelsträngige DNA relativ flexibel ist, könnte das
Flavin auch reduziert werden, in dem es sich der
Elektrodenoberfläche annähert, ohne dass eine
Hybridisierung stattgefunden hat. Dabei ist fraglich,
ob bzw. wie beide Situationen (Flavinreduktion durch
Annäherung an die Elektrodenoberfläche oder durch ET
entlang der Basenpaare von ds-DNA nach vollständiger
Hybridisierung) unterschieden werden können. Eine
Alternative bietet der in (B) gezeigte Sensoraufbau,
der aus einem adsorbierten PNA-Einzelstrang mit 10
Basen besteht (1). Erfolgt eine Hybridisierung mit der
zu analysierenden DNA mit 20 Basen (2), wird im
nächsten Schritt ein flavinmodifizierter
PNA-Einzelstrang mit 10 Basen zugegeben. Nur wenn auch
dieser eine komplementäre Basensequenz aufweist, ist ET
möglich (3). In diesem Fall sollte es wiederum möglich
sein, Apododecin anzubinden (4) und reduktiv
freizusetzen. Die oben skizzierten Sensoren können noch
dahingehend erweitert werden, dass nach Anbindung von
Apododecin ein fluoreszenzfarbstoffmarkierter
Flavinligand zugegeben und durch eine der weiteren
Flavinbindungstaschen von Dodecin gebunden wird. In
diesem Fall könnte die Anbindung und Freisetzung von
Apododecin auch durch
Oberflächenplasmonen-Fluoreszenzspektroskopie (SPFS)
nachgewiesen werden. Sobald eine Konfiguration gefunden
wird, in der effektiver ET durch DNA-Monolagen
beobachtet werden kann und abgesichert ist, dass dieser
ET entlang der Stapel von komplementären Basen-paaren
abläuft, soll an der gezielten Aufspürung von einzelnen
DNA-Fehlpaarungen gearbeitet werden.